13 KiB
Visualisoi jakaumat
 |
|---|
| Jakaumien visualisointi - Sketchnote by @nitya |
Edellisessä oppitunnissa opit mielenkiintoisia asioita Minnesotan lintuihin liittyvästä datasetistä. Löysit virheellistä dataa visualisoimalla poikkeamia ja tarkastelit lintukategorioiden eroja niiden maksimipituuden perusteella.
Ennakkokysely
Tutki lintudatasettiä
Yksi tapa tutkia dataa on tarkastella sen jakaumaa, eli miten data on järjestetty akselilla. Ehkä haluaisit esimerkiksi oppia datasetin yleisestä jakaumasta Minnesotan lintujen maksimisiipivälin tai maksimikehon massan osalta.
Tutkitaanpa joitakin faktoja datasetin datan jakaumista. Tämän oppituntikansion juurihakemistossa olevassa notebook.ipynb-tiedostossa tuo Pandas, Matplotlib ja datasi:
import pandas as pd
import matplotlib.pyplot as plt
birds = pd.read_csv('../../data/birds.csv')
birds.head()
| Nimi | Tieteellinen nimi | Kategoria | Lahko | Heimo | Suku | Suojelustatus | MinPituus | MaxPituus | MinKehonMassa | MaxKehonMassa | MinSiipiväli | MaxSiipiväli | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Mustavatsainen viheltävä ankka | Dendrocygna autumnalis | Ankat/hanhet/vesilinnut | Anseriformes | Anatidae | Dendrocygna | LC | 47 | 56 | 652 | 1020 | 76 | 94 |
| 1 | Ruostevatsainen viheltävä ankka | Dendrocygna bicolor | Ankat/hanhet/vesilinnut | Anseriformes | Anatidae | Dendrocygna | LC | 45 | 53 | 712 | 1050 | 85 | 93 |
| 2 | Lumihanhi | Anser caerulescens | Ankat/hanhet/vesilinnut | Anseriformes | Anatidae | Anser | LC | 64 | 79 | 2050 | 4050 | 135 | 165 |
| 3 | Rossin hanhi | Anser rossii | Ankat/hanhet/vesilinnut | Anseriformes | Anatidae | Anser | LC | 57.3 | 64 | 1066 | 1567 | 113 | 116 |
| 4 | Iso valkoposkihanhi | Anser albifrons | Ankat/hanhet/vesilinnut | Anseriformes | Anatidae | Anser | LC | 64 | 81 | 1930 | 3310 | 130 | 165 |
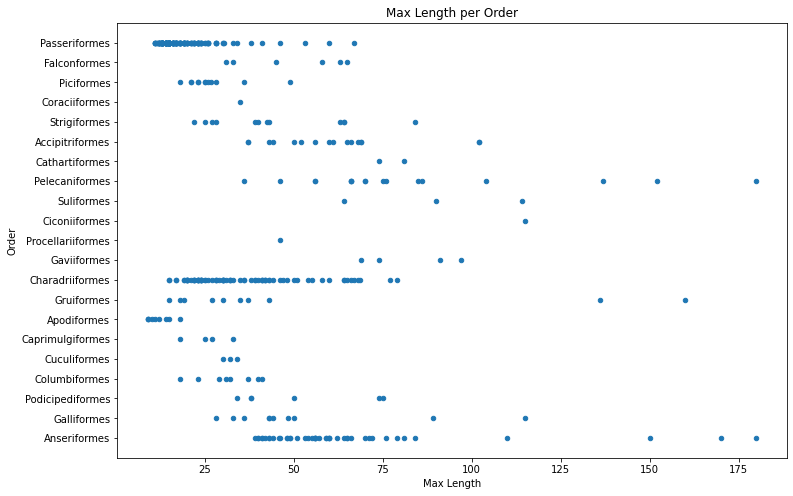
Yleisesti ottaen datan jakaumaa voi nopeasti tarkastella käyttämällä hajontakaaviota, kuten teimme edellisessä oppitunnissa:
birds.plot(kind='scatter',x='MaxLength',y='Order',figsize=(12,8))
plt.title('Max Length per Order')
plt.ylabel('Order')
plt.xlabel('Max Length')
plt.show()
Tämä antaa yleiskuvan lintujen kehon pituuden jakaumasta per lahko, mutta se ei ole optimaalinen tapa esittää todellisia jakaumia. Tämä tehtävä hoidetaan yleensä histogrammin avulla.
Työskentely histogrammien kanssa
Matplotlib tarjoaa erinomaisia tapoja visualisoida datan jakaumia histogrammien avulla. Tämä kaaviotyyppi muistuttaa pylväsdiagrammia, jossa jakauma näkyy pylväiden nousuina ja laskuina. Histogrammin luomiseen tarvitaan numeerista dataa. Histogrammin luomiseksi voit piirtää kaavion määrittämällä tyypiksi 'hist' histogrammia varten. Tämä kaavio näyttää MaxBodyMass-jakauman koko datasetin numeerisen datan osalta. Jakamalla sille annettu data pienempiin osiin (bins), se voi näyttää datan arvojen jakauman:
birds['MaxBodyMass'].plot(kind = 'hist', bins = 10, figsize = (12,12))
plt.show()
Kuten näet, suurin osa datasetin yli 400 linnusta sijoittuu alle 2000 Max Body Mass -arvon alueelle. Saat lisää tietoa datasta muuttamalla bins-parametrin suuremmaksi, esimerkiksi 30:
birds['MaxBodyMass'].plot(kind = 'hist', bins = 30, figsize = (12,12))
plt.show()
Tämä kaavio näyttää jakauman hieman tarkemmalla tasolla. Vähemmän vasemmalle vinoutunut kaavio voitaisiin luoda varmistamalla, että valitset vain datan tietyltä alueelta:
Suodata datasi saadaksesi vain ne linnut, joiden kehon massa on alle 60, ja näytä 40 bins:
filteredBirds = birds[(birds['MaxBodyMass'] > 1) & (birds['MaxBodyMass'] < 60)]
filteredBirds['MaxBodyMass'].plot(kind = 'hist',bins = 40,figsize = (12,12))
plt.show()
✅ Kokeile joitakin muita suodattimia ja datapisteitä. Näyttääksesi datan koko jakauman, poista ['MaxBodyMass']-suodatin ja näytä nimikoidut jakaumat.
Histogrammi tarjoaa myös mukavia väritys- ja nimikointiparannuksia, joita kannattaa kokeilla:
Luo 2D-histogrammi vertaillaksesi kahden jakauman välistä suhdetta. Verrataan MaxBodyMass ja MaxLength. Matplotlib tarjoaa sisäänrakennetun tavan näyttää yhtymäkohdat kirkkaampien värien avulla:
x = filteredBirds['MaxBodyMass']
y = filteredBirds['MaxLength']
fig, ax = plt.subplots(tight_layout=True)
hist = ax.hist2d(x, y)
Näyttää siltä, että näiden kahden elementin välillä on odotettu korrelaatio odotetulla akselilla, ja yksi erityisen vahva yhtymäkohta:
Histogrammit toimivat oletuksena hyvin numeerisen datan kanssa. Entä jos haluat nähdä jakaumia tekstidatan perusteella?
Tutki datasettiä jakaumien osalta tekstidatan avulla
Tämä datasetti sisältää myös hyvää tietoa lintujen kategoriasta, suvusta, lajista ja heimosta sekä niiden suojelustatuksesta. Tutkitaanpa tätä suojelustatustietoa. Mikä on lintujen jakauma niiden suojelustatuksen mukaan?
✅ Datasetissä käytetään useita lyhenteitä kuvaamaan suojelustatusta. Nämä lyhenteet tulevat IUCN:n punaisen listan kategorioista, organisaatiosta, joka luokittelee lajien statuksen.
- CR: Äärimmäisen uhanalainen
- EN: Uhanalainen
- EX: Sukupuuttoon kuollut
- LC: Elinvoimainen
- NT: Lähes uhanalainen
- VU: Haavoittuvainen
Nämä ovat tekstipohjaisia arvoja, joten sinun täytyy tehdä muunnos histogrammin luomiseksi. Käytä filteredBirds-dataframea ja näytä sen suojelustatus yhdessä minimisiipivälin kanssa. Mitä huomaat?
x1 = filteredBirds.loc[filteredBirds.ConservationStatus=='EX', 'MinWingspan']
x2 = filteredBirds.loc[filteredBirds.ConservationStatus=='CR', 'MinWingspan']
x3 = filteredBirds.loc[filteredBirds.ConservationStatus=='EN', 'MinWingspan']
x4 = filteredBirds.loc[filteredBirds.ConservationStatus=='NT', 'MinWingspan']
x5 = filteredBirds.loc[filteredBirds.ConservationStatus=='VU', 'MinWingspan']
x6 = filteredBirds.loc[filteredBirds.ConservationStatus=='LC', 'MinWingspan']
kwargs = dict(alpha=0.5, bins=20)
plt.hist(x1, **kwargs, color='red', label='Extinct')
plt.hist(x2, **kwargs, color='orange', label='Critically Endangered')
plt.hist(x3, **kwargs, color='yellow', label='Endangered')
plt.hist(x4, **kwargs, color='green', label='Near Threatened')
plt.hist(x5, **kwargs, color='blue', label='Vulnerable')
plt.hist(x6, **kwargs, color='gray', label='Least Concern')
plt.gca().set(title='Conservation Status', ylabel='Min Wingspan')
plt.legend();
Näyttää siltä, ettei minimisiipivälin ja suojelustatuksen välillä ole hyvää korrelaatiota. Testaa datasetin muita elementtejä tällä menetelmällä. Voit kokeilla myös erilaisia suodattimia. Löydätkö mitään korrelaatiota?
Tiheyskaaviot
Olet ehkä huomannut, että tähän mennessä tarkastellut histogrammit ovat "askelmia" eivätkä virtaa sujuvasti kaaren muodossa. Näyttääksesi tasaisemman tiheyskaavion, voit kokeilla tiheyskaaviota.
Tiheyskaavioiden kanssa työskennellessäsi tutustu uuteen kaaviokirjastoon, Seaborn.
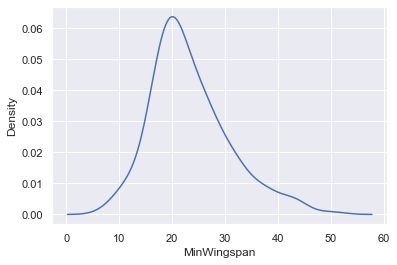
Lataa Seaborn ja kokeile perus tiheyskaaviota:
import seaborn as sns
import matplotlib.pyplot as plt
sns.kdeplot(filteredBirds['MinWingspan'])
plt.show()
Voit nähdä, kuinka kaavio muistuttaa aiempaa minimisiipivälin kaaviota; se on vain hieman tasaisempi. Seabornin dokumentaation mukaan "histogrammiin verrattuna KDE voi tuottaa kaavion, joka on vähemmän sekava ja helpommin tulkittava, erityisesti piirtäessä useita jakaumia. Mutta se voi aiheuttaa vääristymiä, jos taustalla oleva jakauma on rajattu tai ei tasainen. Kuten histogrammi, myös esityksen laatu riippuu hyvien tasoitusparametrien valinnasta." lähde Toisin sanoen, poikkeamat kuten aina saavat kaaviosi käyttäytymään huonosti.
Jos haluaisit palata siihen epätasaiseen MaxBodyMass-linjaan toisessa rakentamassasi kaaviossa, voisit tasoittaa sen hyvin luomalla sen uudelleen tällä menetelmällä:
sns.kdeplot(filteredBirds['MaxBodyMass'])
plt.show()
Jos haluaisit tasaisen, mutta ei liian tasaisen linjan, muokkaa bw_adjust-parametria:
sns.kdeplot(filteredBirds['MaxBodyMass'], bw_adjust=.2)
plt.show()
✅ Lue tämän kaaviotyypin käytettävissä olevista parametreista ja kokeile!
Tämä kaaviotyyppi tarjoaa kauniisti selittäviä visualisointeja. Muutamalla koodirivillä voit esimerkiksi näyttää lintujen maksimikehon massan tiheyden per lahko:
sns.kdeplot(
data=filteredBirds, x="MaxBodyMass", hue="Order",
fill=True, common_norm=False, palette="crest",
alpha=.5, linewidth=0,
)
Voit myös kartoittaa useiden muuttujien tiheyden yhdessä kaaviossa. Testaa lintujen MaxLength ja MinLength verrattuna niiden suojelustatukseen:
sns.kdeplot(data=filteredBirds, x="MinLength", y="MaxLength", hue="ConservationStatus")
Ehkä kannattaa tutkia, onko 'Haavoittuvaiset' lintujen pituuksien mukaan muodostuva klusteri merkityksellinen vai ei.
🚀 Haaste
Histogrammit ovat kehittyneempiä kaaviotyyppejä kuin perushajontakaaviot, pylväsdiagrammit tai viivakaaviot. Etsi internetistä hyviä esimerkkejä histogrammien käytöstä. Miten niitä käytetään, mitä ne osoittavat ja millä aloilla tai tutkimusalueilla niitä yleensä käytetään?
Jälkikysely
Kertaus ja itseopiskelu
Tässä oppitunnissa käytit Matplotlibia ja aloitit työskentelyn Seabornin kanssa näyttääkseen kehittyneempiä kaavioita. Tutki kdeplot-toimintoa Seabornissa, "jatkuva todennäköisyystiheyskäyrä yhdessä tai useammassa ulottuvuudessa". Lue dokumentaatio ymmärtääksesi, miten se toimii.
Tehtävä
Vastuuvapauslauseke:
Tämä asiakirja on käännetty käyttämällä tekoälypohjaista käännöspalvelua Co-op Translator. Vaikka pyrimme tarkkuuteen, huomioithan, että automaattiset käännökset voivat sisältää virheitä tai epätarkkuuksia. Alkuperäistä asiakirjaa sen alkuperäisellä kielellä tulisi pitää ensisijaisena lähteenä. Kriittisen tiedon osalta suositellaan ammattimaista ihmiskääntämistä. Emme ole vastuussa väärinkäsityksistä tai virhetulkinnoista, jotka johtuvat tämän käännöksen käytöstä.