|
|
4 weeks ago | |
|---|---|---|
| .. | ||
| README.md | 4 weeks ago | |
| assignment.md | 4 weeks ago | |
README.md
Визуализиране на разпределения
 |
|---|
| Визуализиране на разпределения - Скетч от @nitya |
В предишния урок научихте някои интересни факти за набор от данни за птиците в Минесота. Открихте грешни данни чрез визуализиране на отклонения и разгледахте разликите между категориите птици според тяхната максимална дължина.
Тест преди лекцията
Изследване на набора от данни за птиците
Друг начин за анализиране на данни е като се разгледа тяхното разпределение, или как данните са организирани по дадена ос. Например, може би искате да научите за общото разпределение в този набор от данни на максималния размах на крилата или максималната телесна маса на птиците в Минесота.
Нека открием някои факти за разпределенията на данните в този набор. Във файла notebook.ipynb в основната папка на този урок, импортирайте Pandas, Matplotlib и вашите данни:
import pandas as pd
import matplotlib.pyplot as plt
birds = pd.read_csv('../../data/birds.csv')
birds.head()
| Име | Научно име | Категория | Разред | Семейство | Род | Статус на опазване | Мин. дължина | Макс. дължина | Мин. телесна маса | Макс. телесна маса | Мин. размах на крилата | Макс. размах на крилата | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Черно-коремна свиреща патица | Dendrocygna autumnalis | Патици/Гъски/Водоплаващи | Anseriformes | Anatidae | Dendrocygna | LC | 47 | 56 | 652 | 1020 | 76 | 94 |
| 1 | Ръждива свиреща патица | Dendrocygna bicolor | Патици/Гъски/Водоплаващи | Anseriformes | Anatidae | Dendrocygna | LC | 45 | 53 | 712 | 1050 | 85 | 93 |
| 2 | Снежна гъска | Anser caerulescens | Патици/Гъски/Водоплаващи | Anseriformes | Anatidae | Anser | LC | 64 | 79 | 2050 | 4050 | 135 | 165 |
| 3 | Гъска на Рос | Anser rossii | Патици/Гъски/Водоплаващи | Anseriformes | Anatidae | Anser | LC | 57.3 | 64 | 1066 | 1567 | 113 | 116 |
| 4 | Голямо бяло-чело гъска | Anser albifrons | Патици/Гъски/Водоплаващи | Anseriformes | Anatidae | Anser | LC | 64 | 81 | 1930 | 3310 | 130 | 165 |
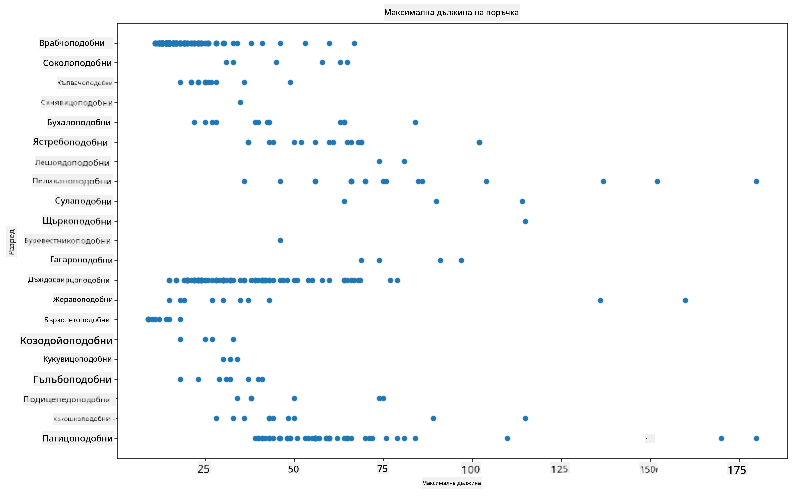
Обикновено можете бързо да разгледате начина, по който данните са разпределени, като използвате точкова диаграма, както направихме в предишния урок:
birds.plot(kind='scatter',x='MaxLength',y='Order',figsize=(12,8))
plt.title('Max Length per Order')
plt.ylabel('Order')
plt.xlabel('Max Length')
plt.show()
Това дава общ преглед на разпределението на дължината на тялото по разред на птиците, но не е оптималният начин за показване на истински разпределения. Тази задача обикновено се изпълнява чрез създаване на хистограма.
Работа с хистограми
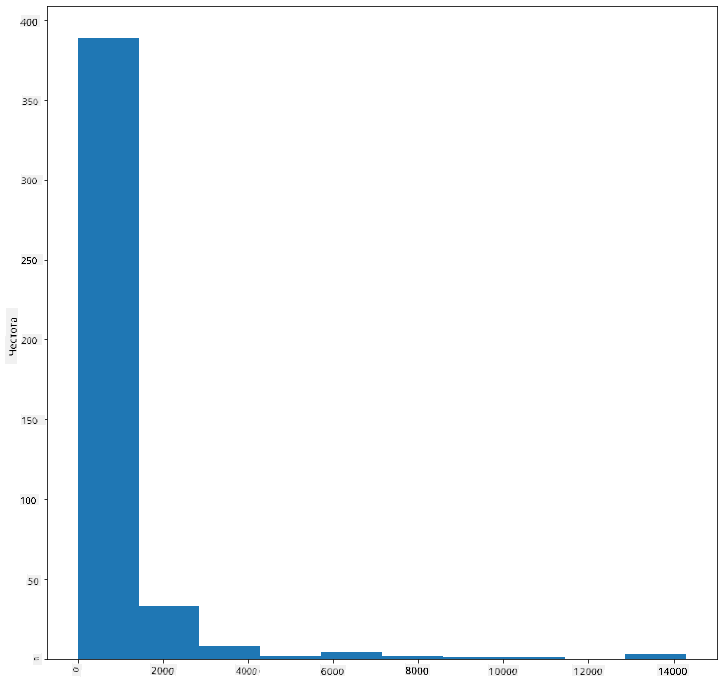
Matplotlib предлага много добри начини за визуализиране на разпределението на данни чрез хистограми. Този тип диаграма е подобен на стълбовидна диаграма, където разпределението може да се види чрез покачването и спадането на стълбовете. За да създадете хистограма, ви трябват числови данни. За да създадете хистограма, можете да начертаете диаграма, като зададете типа като 'hist' за хистограма. Тази диаграма показва разпределението на MaxBodyMass за целия набор от числови данни. Като разделя масива от данни на по-малки групи, тя може да покаже разпределението на стойностите на данните:
birds['MaxBodyMass'].plot(kind = 'hist', bins = 10, figsize = (12,12))
plt.show()
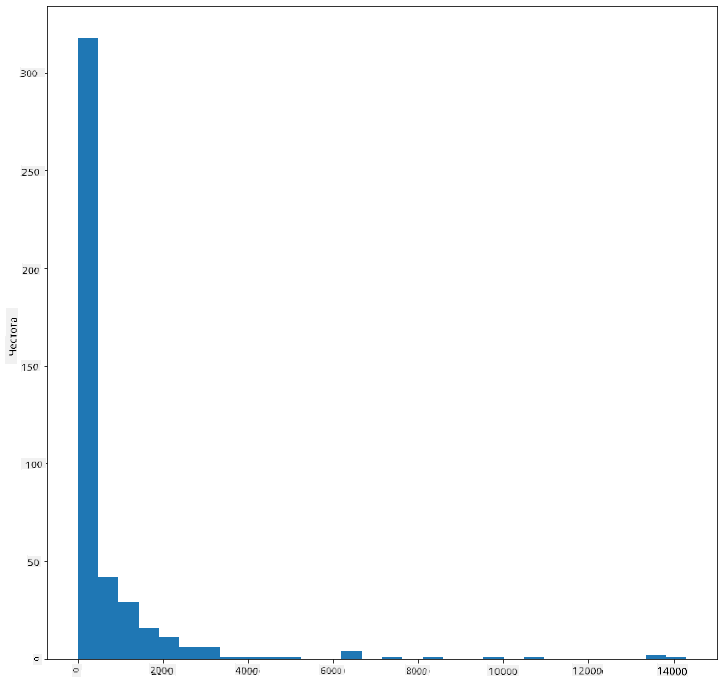
Както виждате, повечето от 400+ птици в този набор от данни попадат в диапазона под 2000 за тяхната максимална телесна маса. Получете повече информация за данните, като промените параметъра bins на по-голямо число, например 30:
birds['MaxBodyMass'].plot(kind = 'hist', bins = 30, figsize = (12,12))
plt.show()
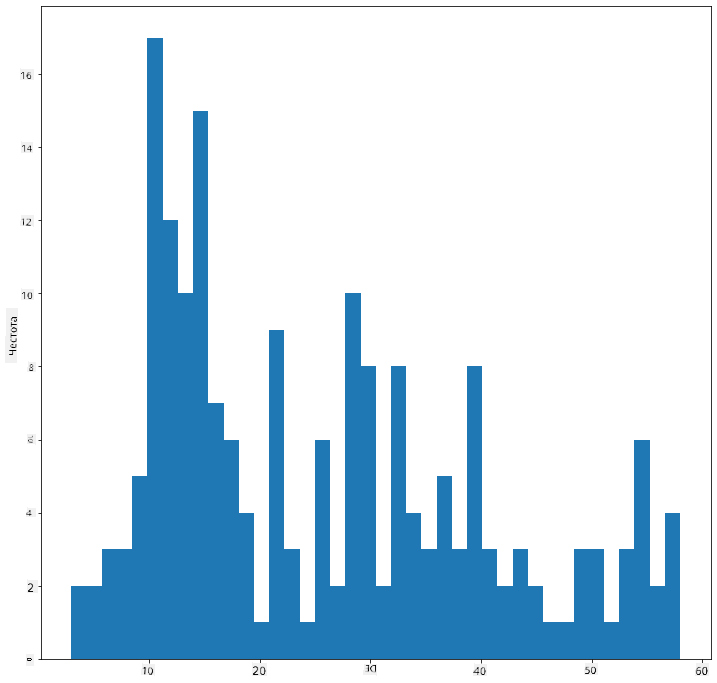
Тази диаграма показва разпределението по малко по-гранулиран начин. Диаграма, която е по-малко изкривена наляво, може да бъде създадена, като се уверите, че избирате само данни в даден диапазон:
Филтрирайте данните си, за да получите само тези птици, чиято телесна маса е под 60, и покажете 40 bins:
filteredBirds = birds[(birds['MaxBodyMass'] > 1) & (birds['MaxBodyMass'] < 60)]
filteredBirds['MaxBodyMass'].plot(kind = 'hist',bins = 40,figsize = (12,12))
plt.show()
✅ Опитайте някои други филтри и точки от данни. За да видите пълното разпределение на данните, премахнете филтъра ['MaxBodyMass'], за да покажете етикетирани разпределения.
Хистограмата предлага някои приятни подобрения в цветовете и етикетирането, които също можете да опитате:
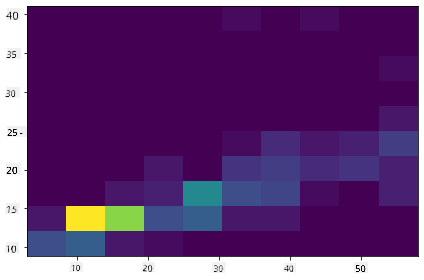
Създайте 2D хистограма, за да сравните връзката между две разпределения. Нека сравним MaxBodyMass срещу MaxLength. Matplotlib предлага вграден начин за показване на сближаване чрез по-ярки цветове:
x = filteredBirds['MaxBodyMass']
y = filteredBirds['MaxLength']
fig, ax = plt.subplots(tight_layout=True)
hist = ax.hist2d(x, y)
Изглежда, че има очаквана корелация между тези два елемента по очаквана ос, с една особено силна точка на сближаване:
Хистограмите работят добре по подразбиране за числови данни. Ами ако трябва да видите разпределения според текстови данни?
Изследване на набора от данни за разпределения с текстови данни
Този набор от данни включва също добра информация за категорията на птиците, техния род, вид и семейство, както и за статуса им на опазване. Нека разгледаме тази информация за опазване. Какво е разпределението на птиците според техния статус на опазване?
✅ В набора от данни се използват няколко съкращения за описание на статуса на опазване. Тези съкращения идват от IUCN Red List Categories, организация, която каталогизира статуса на видовете.
- CR: Критично застрашен
- EN: Застрашен
- EX: Изчезнал
- LC: Най-малка загриженост
- NT: Близо до застрашен
- VU: Уязвим
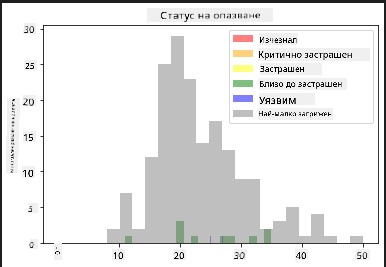
Това са текстови стойности, така че ще трябва да направите трансформация, за да създадете хистограма. Използвайки dataframe-а filteredBirds, покажете статуса на опазване заедно с минималния размах на крилата. Какво виждате?
x1 = filteredBirds.loc[filteredBirds.ConservationStatus=='EX', 'MinWingspan']
x2 = filteredBirds.loc[filteredBirds.ConservationStatus=='CR', 'MinWingspan']
x3 = filteredBirds.loc[filteredBirds.ConservationStatus=='EN', 'MinWingspan']
x4 = filteredBirds.loc[filteredBirds.ConservationStatus=='NT', 'MinWingspan']
x5 = filteredBirds.loc[filteredBirds.ConservationStatus=='VU', 'MinWingspan']
x6 = filteredBirds.loc[filteredBirds.ConservationStatus=='LC', 'MinWingspan']
kwargs = dict(alpha=0.5, bins=20)
plt.hist(x1, **kwargs, color='red', label='Extinct')
plt.hist(x2, **kwargs, color='orange', label='Critically Endangered')
plt.hist(x3, **kwargs, color='yellow', label='Endangered')
plt.hist(x4, **kwargs, color='green', label='Near Threatened')
plt.hist(x5, **kwargs, color='blue', label='Vulnerable')
plt.hist(x6, **kwargs, color='gray', label='Least Concern')
plt.gca().set(title='Conservation Status', ylabel='Min Wingspan')
plt.legend();
Не изглежда да има добра корелация между минималния размах на крилата и статуса на опазване. Тествайте други елементи от набора от данни, използвайки този метод. Можете да опитате различни филтри. Откривате ли някаква корелация?
Плътностни диаграми
Може би сте забелязали, че хистограмите, които разгледахме досега, са "стъпаловидни" и не се движат плавно в дъга. За да покажете по-гладка диаграма на плътността, можете да опитате плътностна диаграма.
За да работите с плътностни диаграми, запознайте се с нова библиотека за визуализация, Seaborn.
Зареждайки Seaborn, опитайте основна плътностна диаграма:
import seaborn as sns
import matplotlib.pyplot as plt
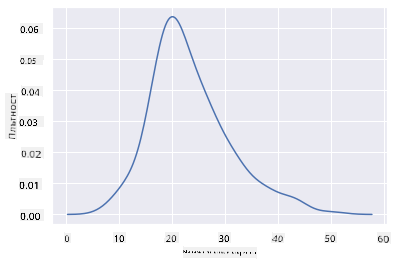
sns.kdeplot(filteredBirds['MinWingspan'])
plt.show()
Можете да видите как диаграмата отразява предишната за данните за минималния размах на крилата; просто е малко по-гладка. Според документацията на Seaborn, "В сравнение с хистограма, KDE може да произведе диаграма, която е по-малко претрупана и по-лесна за интерпретиране, особено когато се чертаят множество разпределения. Но има потенциал да въведе изкривявания, ако основното разпределение е ограничено или не е гладко. Както при хистограмата, качеството на представянето също зависи от избора на добри параметри за изглаждане." източник С други думи, отклоненията, както винаги, ще направят вашите диаграми да се държат странно.
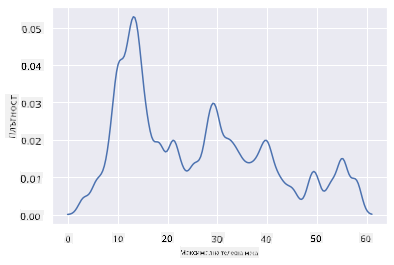
Ако искате да преразгледате тази назъбена линия за MaxBodyMass във втората диаграма, която създадохте, можете да я изгладите много добре, като я пресъздадете с този метод:
sns.kdeplot(filteredBirds['MaxBodyMass'])
plt.show()
Ако искате гладка, но не прекалено гладка линия, редактирайте параметъра bw_adjust:
sns.kdeplot(filteredBirds['MaxBodyMass'], bw_adjust=.2)
plt.show()
✅ Прочетете за наличните параметри за този тип диаграма и експериментирайте!
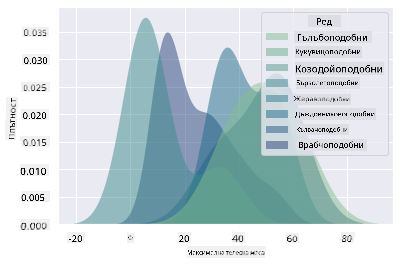
Този тип диаграма предлага красиво обяснителни визуализации. С няколко реда код, например, можете да покажете плътността на максималната телесна маса по разред на птиците:
sns.kdeplot(
data=filteredBirds, x="MaxBodyMass", hue="Order",
fill=True, common_norm=False, palette="crest",
alpha=.5, linewidth=0,
)
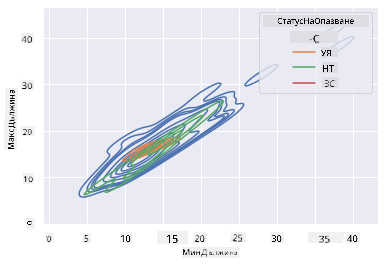
Можете също така да картографирате плътността на няколко променливи в една диаграма. Тествайте максималната и минималната дължина на птица спрямо техния статус на опазване:
sns.kdeplot(data=filteredBirds, x="MinLength", y="MaxLength", hue="ConservationStatus")
Може би си струва да се изследва дали клъстерът на "Уязвимите" птици според техните дължини е значим или не.
🚀 Предизвикателство
Хистограмите са по-сложен тип диаграма от основните точкови диаграми, стълбовидни диаграми или линейни диаграми. Потърсете в интернет добри примери за използването на хистограми. Как се използват, какво демонстрират и в какви области или сфери на изследване обикновено се използват?
Тест след лекцията
Преглед и самостоятелно обучение
В този урок използвахте Matplotlib и започнахте да работите със Seaborn, за да създавате по-сложни диаграми. Направете проучване за kdeplot в Seaborn, "непрекъсната крива на вероятностната плътност в едно или повече измерения". Прочетете документацията, за да разберете как работи.
Задание
Отказ от отговорност:
Този документ е преведен с помощта на AI услуга за превод Co-op Translator. Въпреки че се стремим към точност, моля, имайте предвид, че автоматизираните преводи може да съдържат грешки или неточности. Оригиналният документ на неговия роден език трябва да се счита за авторитетен източник. За критична информация се препоръчва професионален човешки превод. Не носим отговорност за недоразумения или погрешни интерпретации, произтичащи от използването на този превод.