|
|
4 weeks ago | |
|---|---|---|
| .. | ||
| README.md | 4 weeks ago | |
| assignment.md | 4 weeks ago | |
README.md
Visualisierung von Verteilungen
 |
|---|
| Visualisierung von Verteilungen - Sketchnote von @nitya |
In der vorherigen Lektion hast du einige interessante Fakten über einen Datensatz zu den Vögeln von Minnesota gelernt. Du hast fehlerhafte Daten durch die Visualisierung von Ausreißern entdeckt und die Unterschiede zwischen Vogelkategorien anhand ihrer maximalen Länge betrachtet.
Quiz vor der Lektion
Erkunde den Vogeldatensatz
Eine weitere Möglichkeit, Daten zu analysieren, besteht darin, ihre Verteilung zu betrachten, also wie die Daten entlang einer Achse organisiert sind. Vielleicht möchtest du beispielsweise die allgemeine Verteilung der maximalen Flügelspannweite oder der maximalen Körpermasse der Vögel von Minnesota in diesem Datensatz untersuchen.
Lass uns einige Fakten über die Verteilungen der Daten in diesem Datensatz entdecken. Importiere in deiner R-Konsole ggplot2 und die Datenbank. Entferne die Ausreißer aus der Datenbank, wie im vorherigen Thema beschrieben.
library(ggplot2)
birds <- read.csv("../../data/birds.csv",fileEncoding="UTF-8-BOM")
birds_filtered <- subset(birds, MaxWingspan < 500)
head(birds_filtered)
| Name | Wissenschaftlicher Name | Kategorie | Ordnung | Familie | Gattung | Schutzstatus | MinLänge | MaxLänge | MinKörpermasse | MaxKörpermasse | MinFlügelspannweite | MaxFlügelspannweite | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Schwarzbauch-Pfeifgans | Dendrocygna autumnalis | Enten/Gänse/Wasservögel | Anseriformes | Anatidae | Dendrocygna | LC | 47 | 56 | 652 | 1020 | 76 | 94 |
| 1 | Fuchsrote Pfeifgans | Dendrocygna bicolor | Enten/Gänse/Wasservögel | Anseriformes | Anatidae | Dendrocygna | LC | 45 | 53 | 712 | 1050 | 85 | 93 |
| 2 | Schneegans | Anser caerulescens | Enten/Gänse/Wasservögel | Anseriformes | Anatidae | Anser | LC | 64 | 79 | 2050 | 4050 | 135 | 165 |
| 3 | Zwerggans | Anser rossii | Enten/Gänse/Wasservögel | Anseriformes | Anatidae | Anser | LC | 57.3 | 64 | 1066 | 1567 | 113 | 116 |
| 4 | Blässgans | Anser albifrons | Enten/Gänse/Wasservögel | Anseriformes | Anatidae | Anser | LC | 64 | 81 | 1930 | 3310 | 130 | 165 |
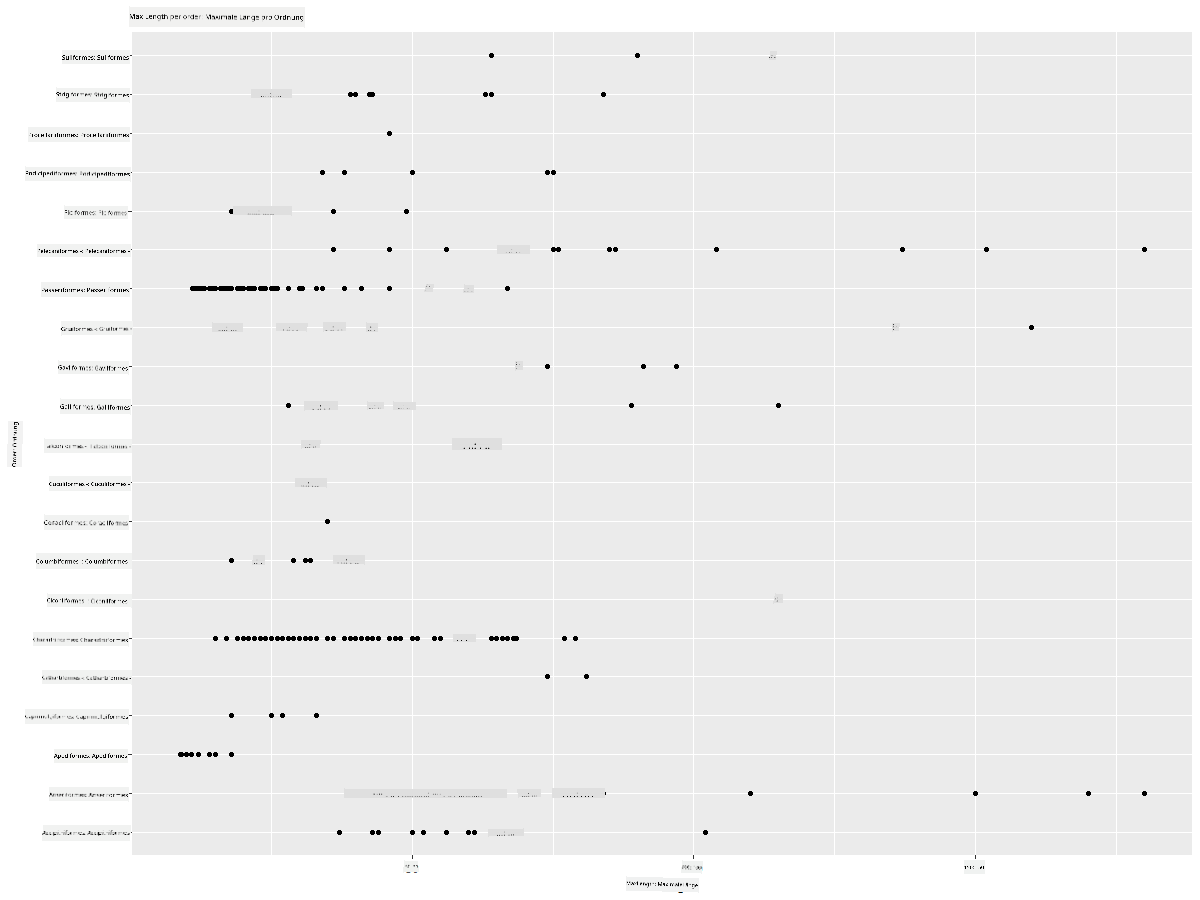
Im Allgemeinen kannst du die Verteilung der Daten schnell mit einem Streudiagramm betrachten, wie wir es in der vorherigen Lektion gemacht haben:
ggplot(data=birds_filtered, aes(x=Order, y=MaxLength,group=1)) +
geom_point() +
ggtitle("Max Length per order") + coord_flip()
Dies gibt einen Überblick über die allgemeine Verteilung der Körperlänge pro Vogelordnung, ist jedoch nicht die optimale Methode, um echte Verteilungen darzustellen. Diese Aufgabe wird normalerweise durch die Erstellung eines Histogramms gelöst.
Arbeiten mit Histogrammen
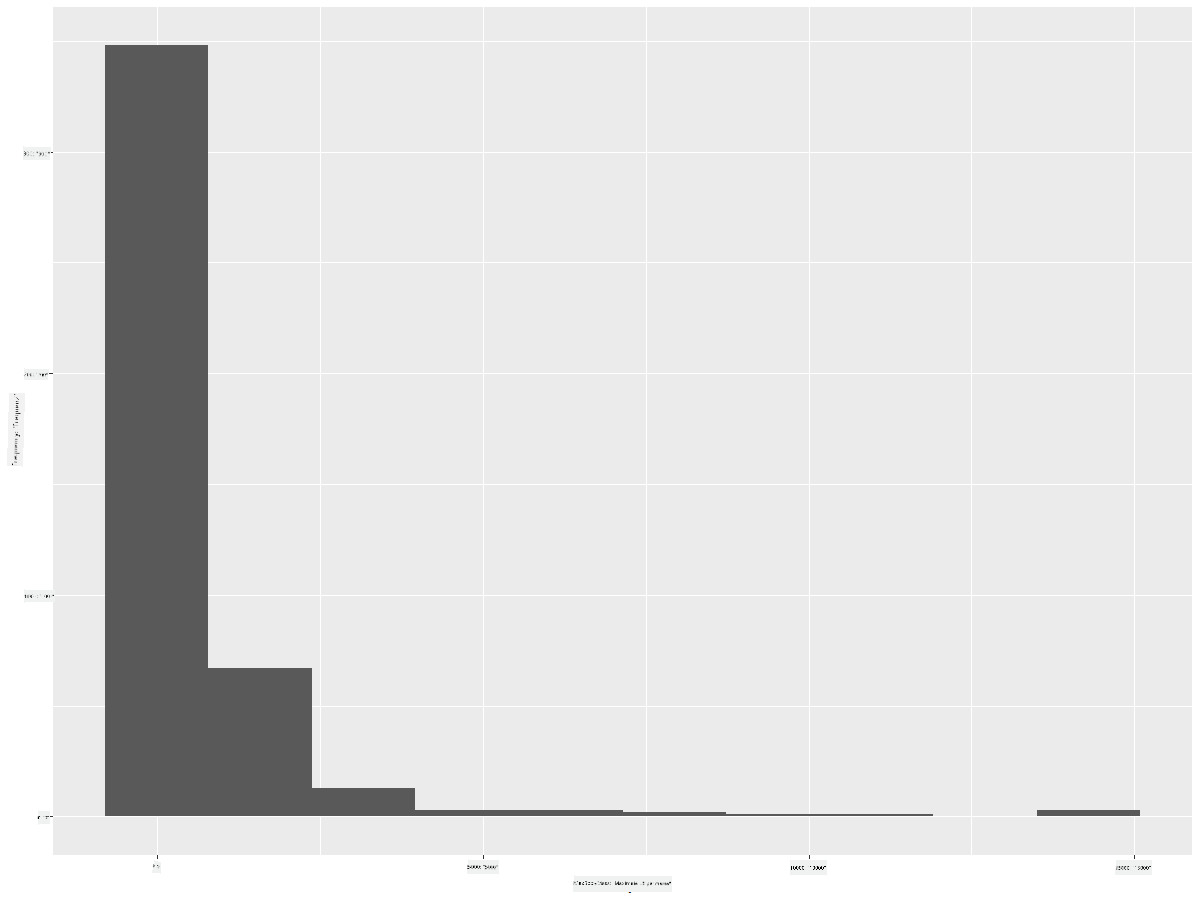
ggplot2 bietet sehr gute Möglichkeiten, die Datenverteilung mit Histogrammen zu visualisieren. Diese Art von Diagramm ähnelt einem Balkendiagramm, bei dem die Verteilung durch das Ansteigen und Abfallen der Balken sichtbar wird. Um ein Histogramm zu erstellen, benötigst du numerische Daten. Um ein Histogramm zu erstellen, kannst du ein Diagramm mit der Art 'hist' für Histogramm zeichnen. Dieses Diagramm zeigt die Verteilung der MaxKörpermasse für den gesamten numerischen Datenbereich des Datensatzes. Indem die Daten in kleinere Intervalle unterteilt werden, kann die Verteilung der Werte dargestellt werden:
ggplot(data = birds_filtered, aes(x = MaxBodyMass)) +
geom_histogram(bins=10)+ylab('Frequency')
Wie du sehen kannst, fallen die meisten der über 400 Vögel in diesem Datensatz in den Bereich unter 2000 für ihre maximale Körpermasse. Erhalte mehr Einblicke in die Daten, indem du den bins-Parameter auf eine höhere Zahl, z. B. 30, änderst:
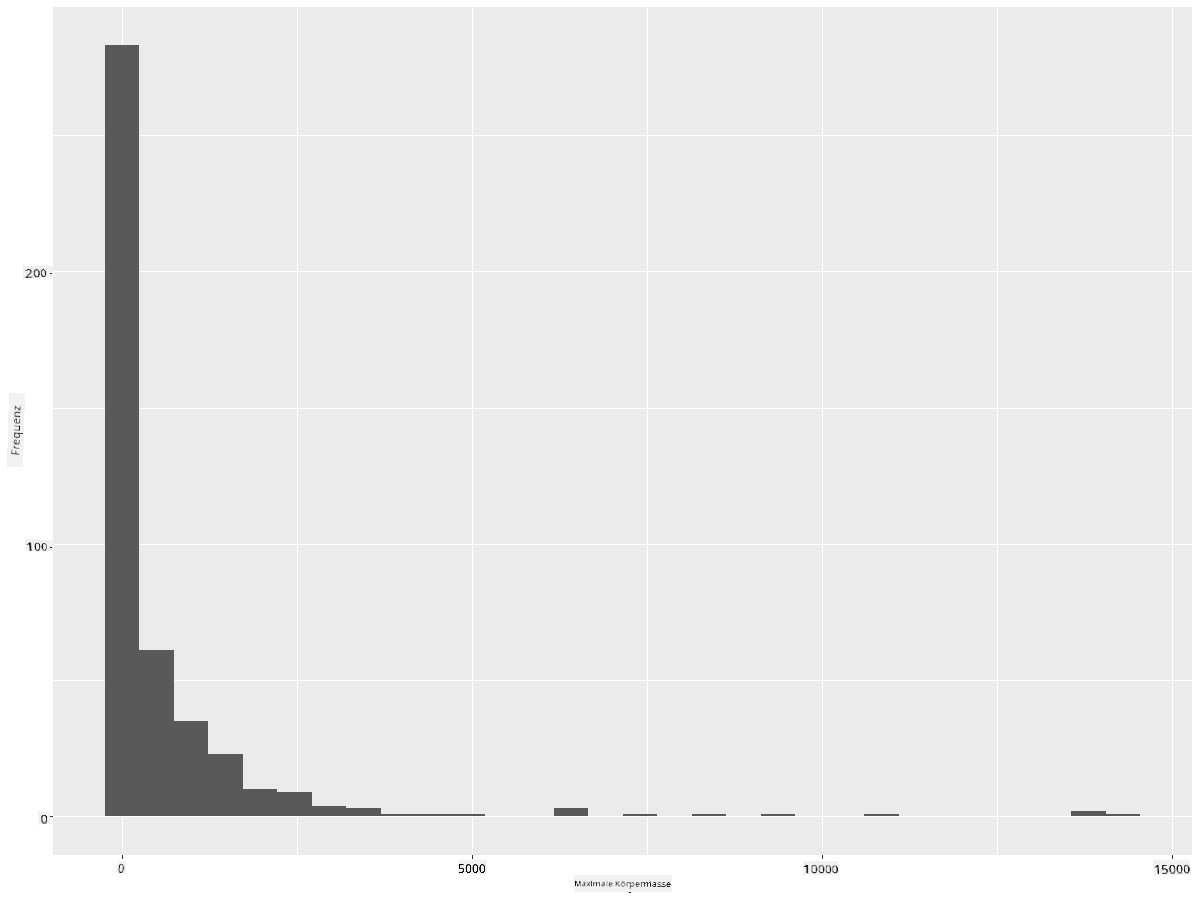
ggplot(data = birds_filtered, aes(x = MaxBodyMass)) + geom_histogram(bins=30)+ylab('Frequency')
Dieses Diagramm zeigt die Verteilung etwas detaillierter. Ein weniger nach links verzerrtes Diagramm könnte erstellt werden, indem du sicherstellst, dass du nur Daten innerhalb eines bestimmten Bereichs auswählst:
Filtere deine Daten, um nur die Vögel zu erhalten, deren Körpermasse unter 60 liegt, und zeige 30 bins:
birds_filtered_1 <- subset(birds_filtered, MaxBodyMass > 1 & MaxBodyMass < 60)
ggplot(data = birds_filtered_1, aes(x = MaxBodyMass)) +
geom_histogram(bins=30)+ylab('Frequency')
✅ Probiere einige andere Filter und Datenpunkte aus. Um die vollständige Verteilung der Daten zu sehen, entferne den ['MaxBodyMass']-Filter, um beschriftete Verteilungen anzuzeigen.
Das Histogramm bietet auch einige schöne Farb- und Beschriftungsoptionen, die du ausprobieren kannst:
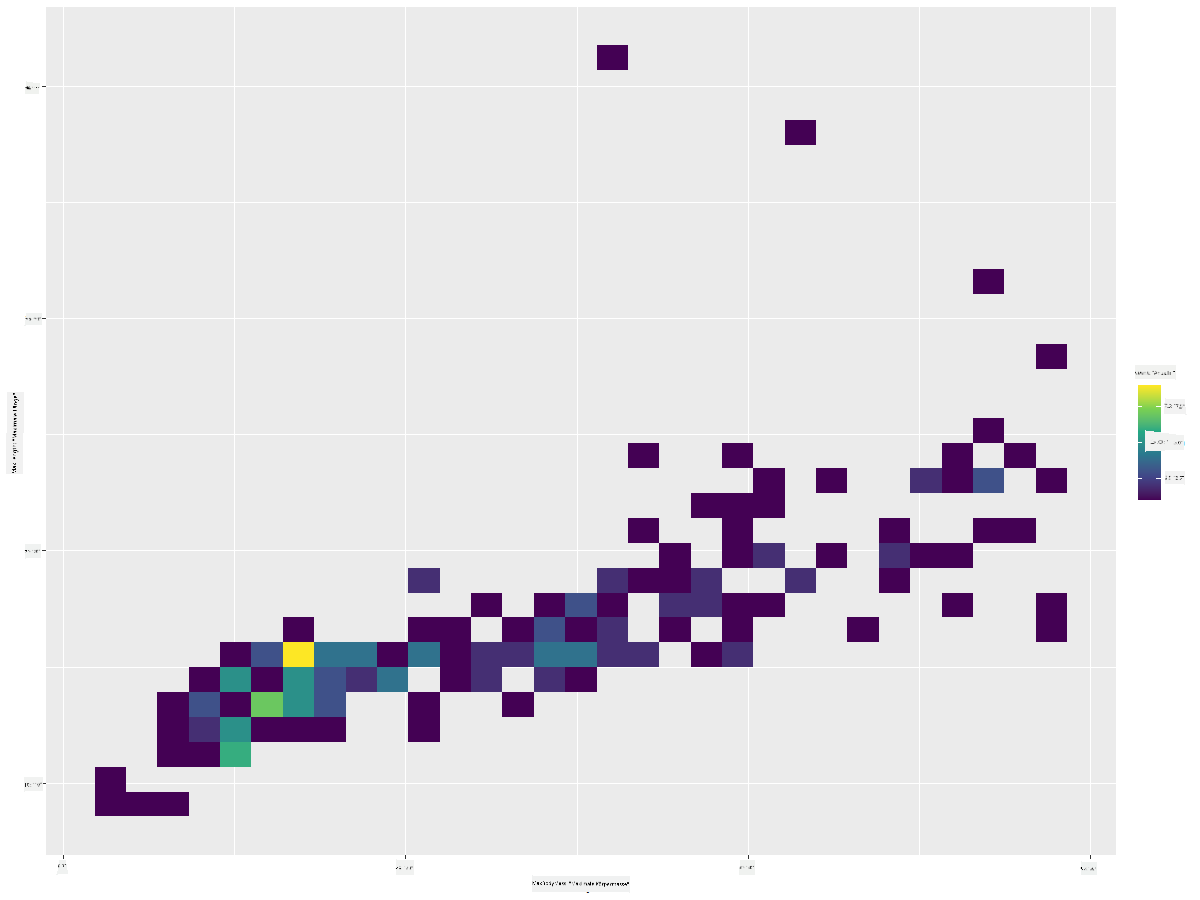
Erstelle ein 2D-Histogramm, um die Beziehung zwischen zwei Verteilungen zu vergleichen. Lass uns MaxBodyMass mit MaxLength vergleichen. ggplot2 bietet eine integrierte Möglichkeit, Konvergenzen mit helleren Farben darzustellen:
ggplot(data=birds_filtered_1, aes(x=MaxBodyMass, y=MaxLength) ) +
geom_bin2d() +scale_fill_continuous(type = "viridis")
Es scheint eine erwartete Korrelation zwischen diesen beiden Elementen entlang einer erwarteten Achse zu geben, mit einem besonders starken Konvergenzpunkt:
Histogramme funktionieren standardmäßig gut für numerische Daten. Was ist, wenn du Verteilungen basierend auf Textdaten sehen möchtest?
Erkunde den Datensatz nach Verteilungen mit Textdaten
Dieser Datensatz enthält auch gute Informationen über die Vogelkategorie sowie deren Gattung, Art und Familie sowie deren Schutzstatus. Lass uns diese Schutzstatus-Informationen genauer betrachten. Wie ist die Verteilung der Vögel nach ihrem Schutzstatus?
✅ Im Datensatz werden mehrere Abkürzungen verwendet, um den Schutzstatus zu beschreiben. Diese Abkürzungen stammen von den IUCN Red List Categories, einer Organisation, die den Status von Arten katalogisiert.
- CR: Kritisch gefährdet
- EN: Gefährdet
- EX: Ausgestorben
- LC: Geringste Sorge
- NT: Potenziell gefährdet
- VU: Verletzlich
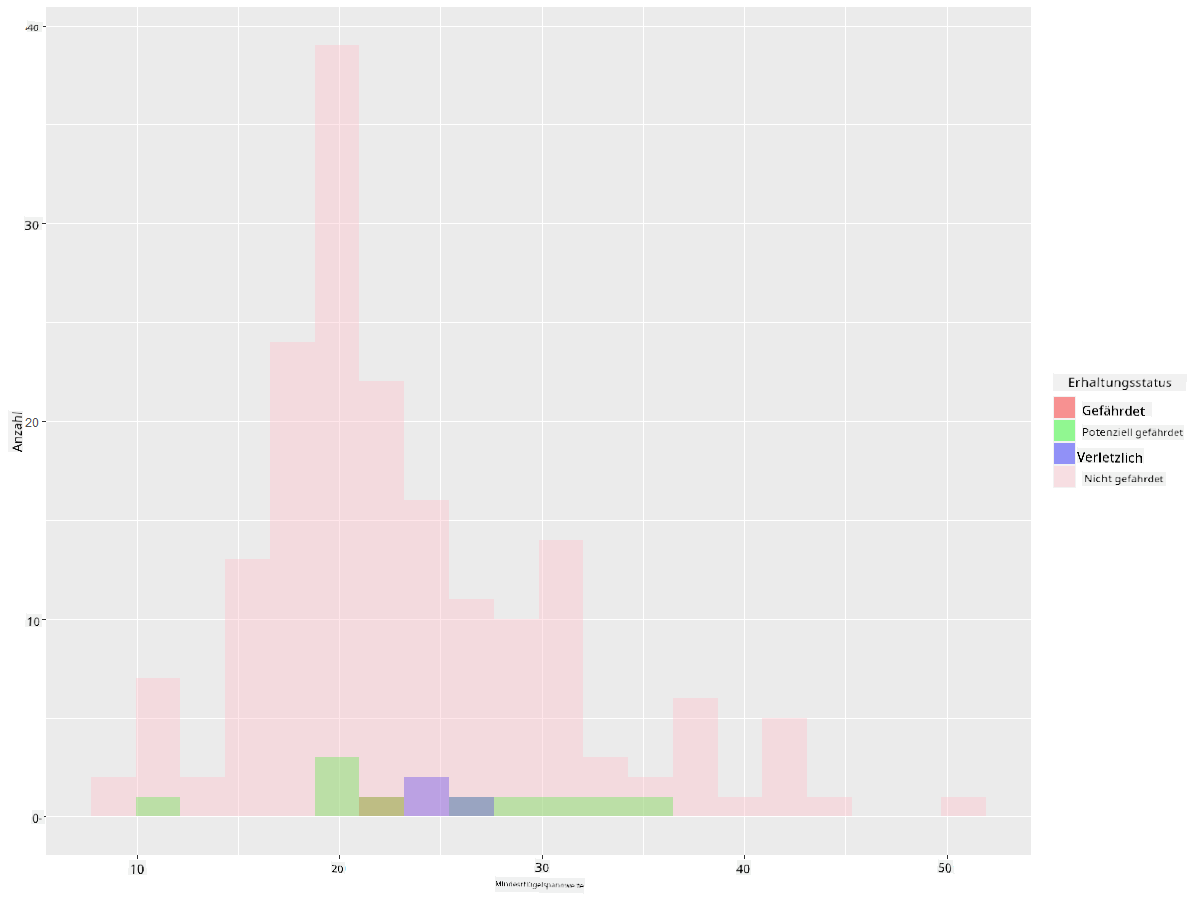
Da es sich um textbasierte Werte handelt, musst du eine Transformation durchführen, um ein Histogramm zu erstellen. Verwende den filteredBirds-Dataframe, um den Schutzstatus zusammen mit der minimalen Flügelspannweite anzuzeigen. Was siehst du?
birds_filtered_1$ConservationStatus[birds_filtered_1$ConservationStatus == 'EX'] <- 'x1'
birds_filtered_1$ConservationStatus[birds_filtered_1$ConservationStatus == 'CR'] <- 'x2'
birds_filtered_1$ConservationStatus[birds_filtered_1$ConservationStatus == 'EN'] <- 'x3'
birds_filtered_1$ConservationStatus[birds_filtered_1$ConservationStatus == 'NT'] <- 'x4'
birds_filtered_1$ConservationStatus[birds_filtered_1$ConservationStatus == 'VU'] <- 'x5'
birds_filtered_1$ConservationStatus[birds_filtered_1$ConservationStatus == 'LC'] <- 'x6'
ggplot(data=birds_filtered_1, aes(x = MinWingspan, fill = ConservationStatus)) +
geom_histogram(position = "identity", alpha = 0.4, bins = 20) +
scale_fill_manual(name="Conservation Status",values=c("red","green","blue","pink"),labels=c("Endangered","Near Threathened","Vulnerable","Least Concern"))
Es scheint keine gute Korrelation zwischen minimaler Flügelspannweite und Schutzstatus zu geben. Teste andere Elemente des Datensatzes mit dieser Methode. Kannst du eine Korrelation finden?
Dichteplots
Vielleicht ist dir aufgefallen, dass die Histogramme, die wir bisher betrachtet haben, 'stufig' sind und nicht fließend in einem Bogen verlaufen. Um ein glatteres Dichtediagramm zu zeigen, kannst du einen Dichteplot ausprobieren.
Lass uns jetzt mit Dichteplots arbeiten!
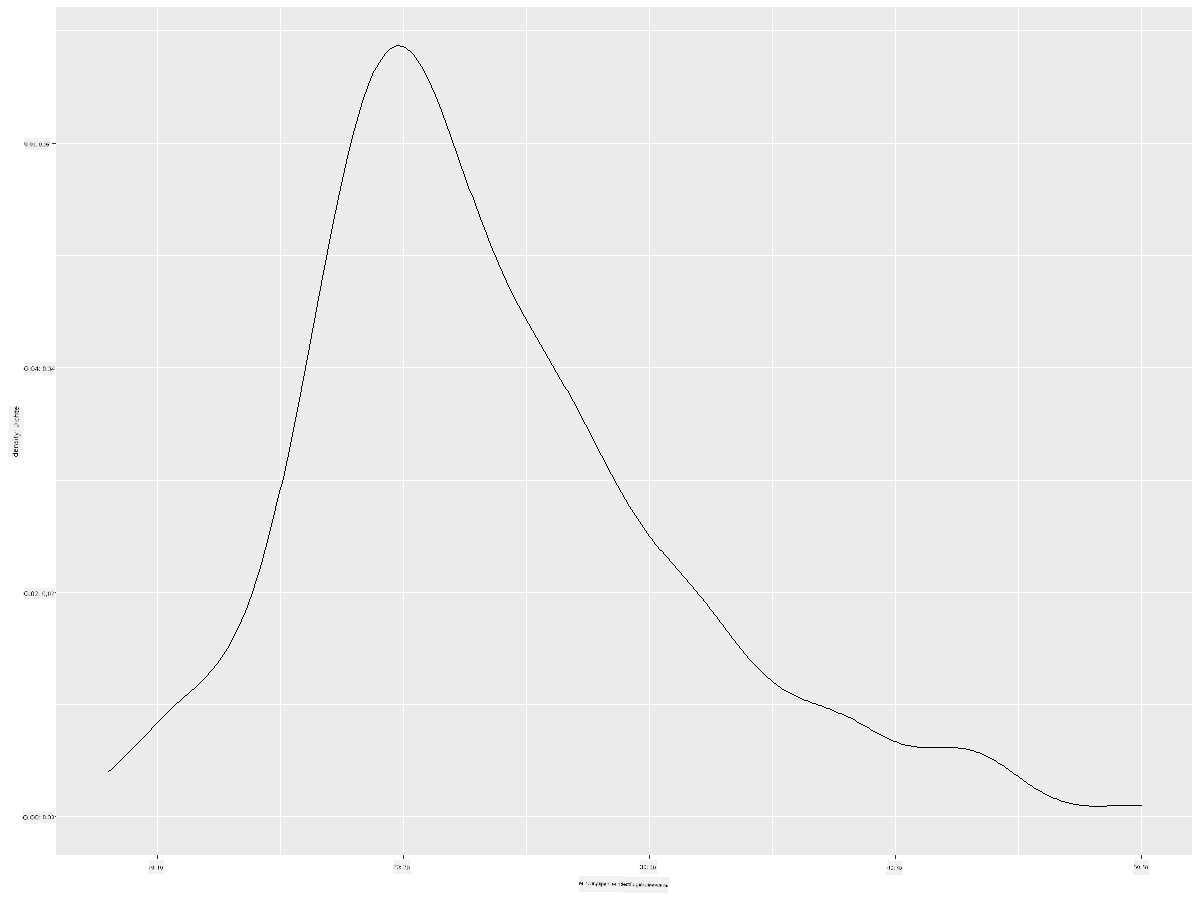
ggplot(data = birds_filtered_1, aes(x = MinWingspan)) +
geom_density()
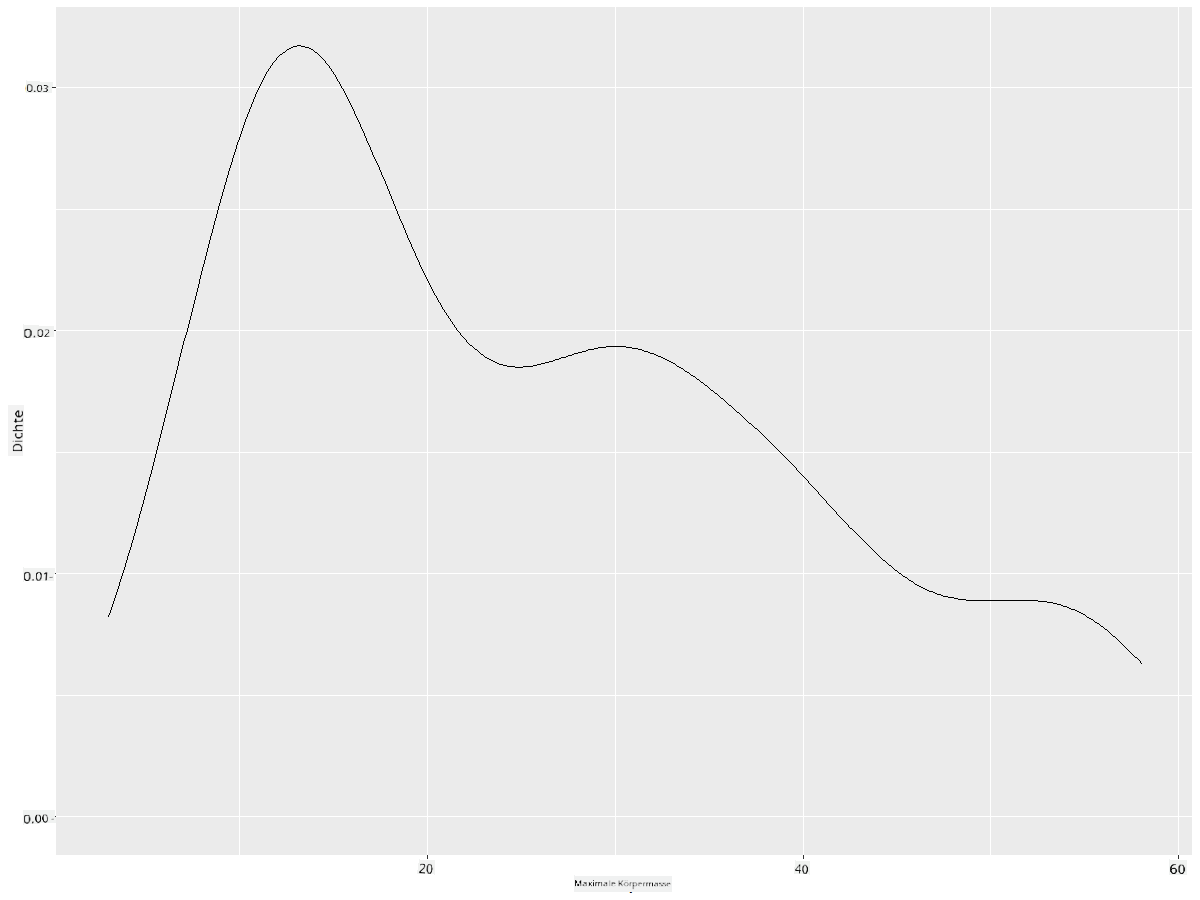
Du kannst sehen, wie der Plot das vorherige Diagramm für die minimale Flügelspannweite widerspiegelt; es ist nur etwas glatter. Wenn du die gezackte MaxKörpermasse-Linie im zweiten Diagramm, das du erstellt hast, glätten möchtest, könntest du dies sehr gut mit dieser Methode tun:
ggplot(data = birds_filtered_1, aes(x = MaxBodyMass)) +
geom_density()
Wenn du eine glatte, aber nicht zu glatte Linie möchtest, bearbeite den adjust-Parameter:
ggplot(data = birds_filtered_1, aes(x = MaxBodyMass)) +
geom_density(adjust = 1/5)
✅ Lies über die verfügbaren Parameter für diesen Diagrammtyp und experimentiere!
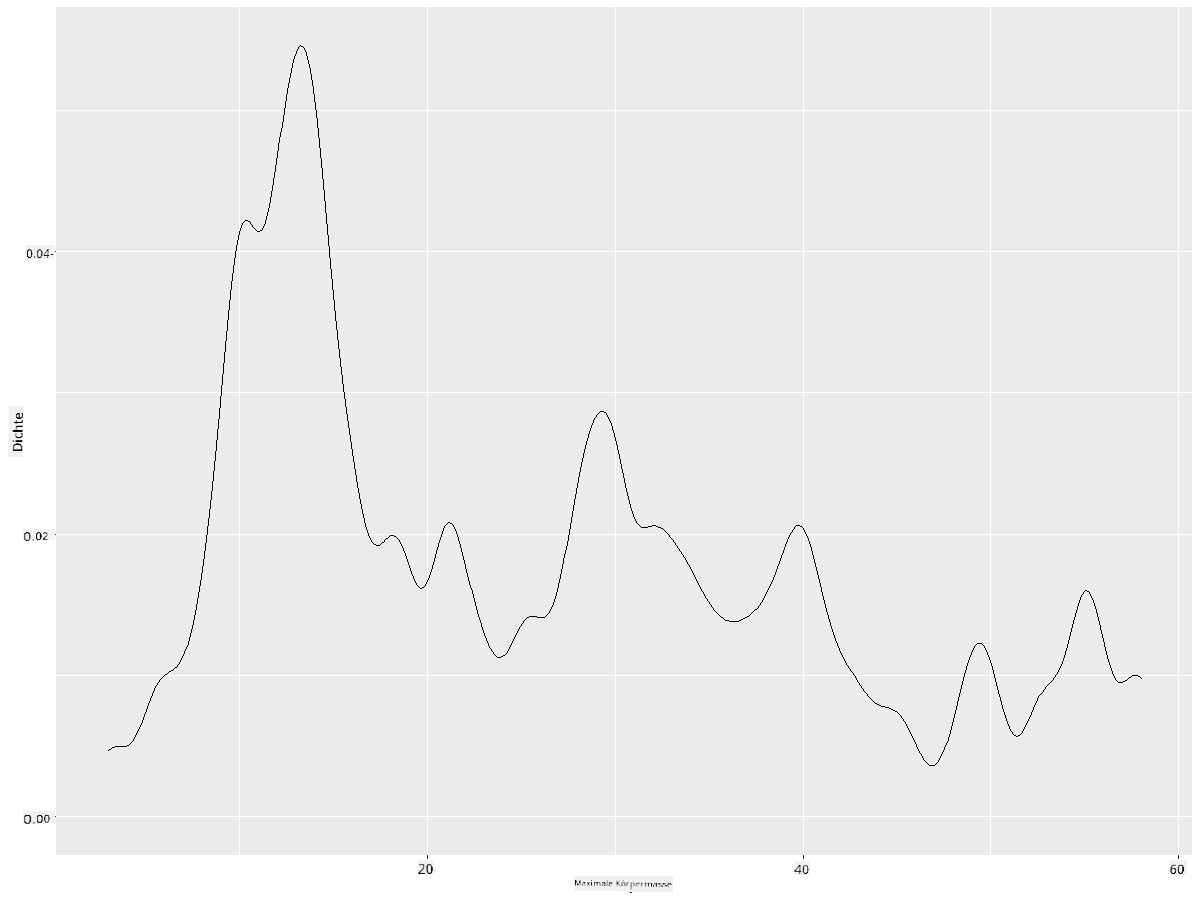
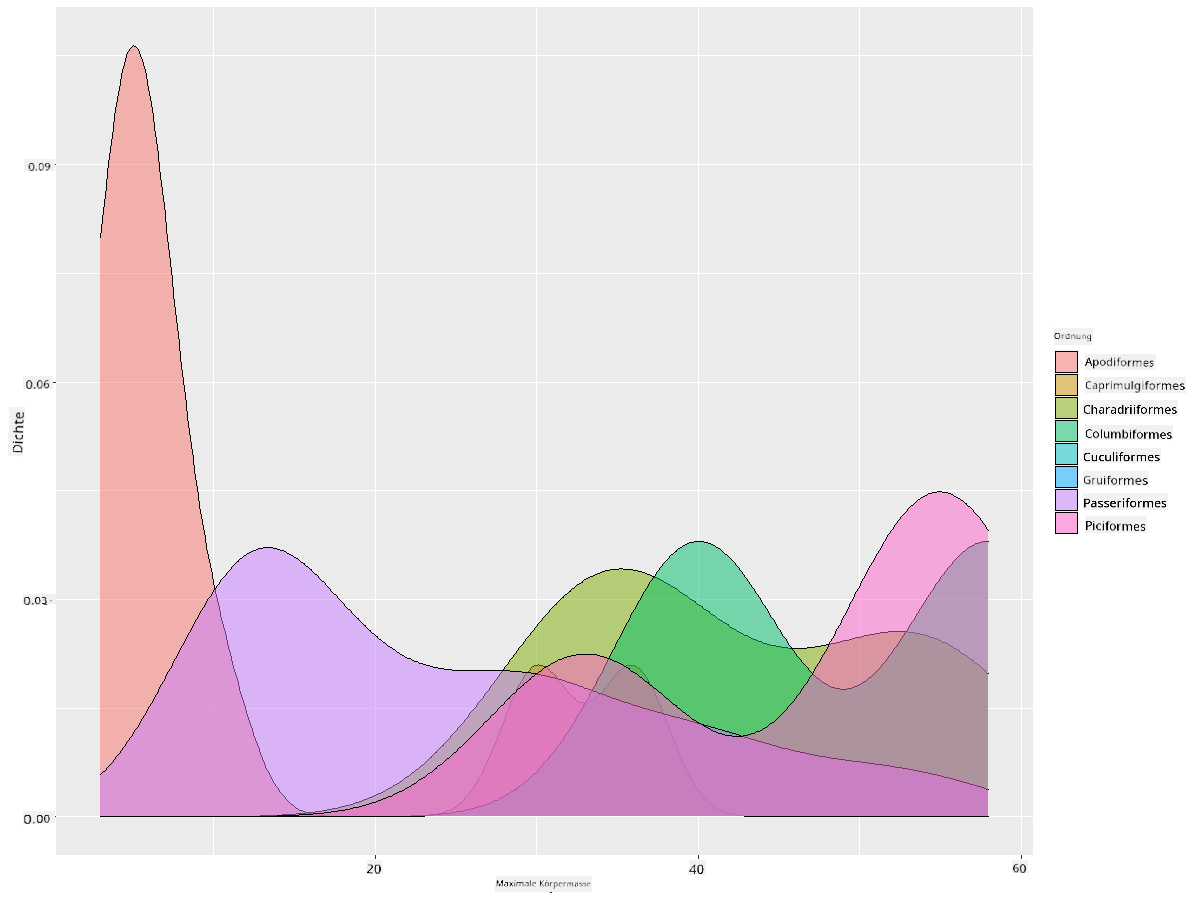
Diese Art von Diagramm bietet wunderschön erklärende Visualisierungen. Mit nur wenigen Codezeilen kannst du beispielsweise die maximale Körpermassendichte pro Vogelordnung anzeigen:
ggplot(data=birds_filtered_1,aes(x = MaxBodyMass, fill = Order)) +
geom_density(alpha=0.5)
🚀 Herausforderung
Histogramme sind eine anspruchsvollere Art von Diagrammen als einfache Streudiagramme, Balkendiagramme oder Liniendiagramme. Suche im Internet nach guten Beispielen für die Verwendung von Histogrammen. Wie werden sie verwendet, was zeigen sie und in welchen Bereichen oder Forschungsfeldern werden sie häufig eingesetzt?
Quiz nach der Lektion
Rückblick & Selbststudium
In dieser Lektion hast du ggplot2 verwendet und begonnen, anspruchsvollere Diagramme zu erstellen. Recherchiere zu geom_density_2d(), einer "kontinuierlichen Wahrscheinlichkeitsdichtekurve in einer oder mehreren Dimensionen". Lies die Dokumentation, um zu verstehen, wie es funktioniert.
Aufgabe
Haftungsausschluss:
Dieses Dokument wurde mit dem KI-Übersetzungsdienst Co-op Translator übersetzt. Obwohl wir uns um Genauigkeit bemühen, beachten Sie bitte, dass automatisierte Übersetzungen Fehler oder Ungenauigkeiten enthalten können. Das Originaldokument in seiner ursprünglichen Sprache sollte als maßgebliche Quelle betrachtet werden. Für kritische Informationen wird eine professionelle menschliche Übersetzung empfohlen. Wir übernehmen keine Haftung für Missverständnisse oder Fehlinterpretationen, die sich aus der Nutzung dieser Übersetzung ergeben.